- 数据库
- 免疫学
- ensembl
- GEO
- biojupies(开源)
- ICGC
- cBioportal(开源)
- TCGA
- GTEx
- UCSC xena
- recount2
- broadinstitute
- Ualcan
- oncolnc
- kmplot
- TRGAted(开源)
- TANRIC
- OncomiR
- CCLE
- HPA
- TCPA
- Oncomine
- Gepia
- MethHC
- LinkedOmics
- CancerSEA
- TSVDB(开源)
数据库
免疫学
ensembl
GEO
biojupies(开源)
- 源码
- 差异基因表达分析
ICGC
cBioportal(开源)
TCGA
GTEx
UCSC xena
- downlaod
- TCGA和GTEx数据合并的项目
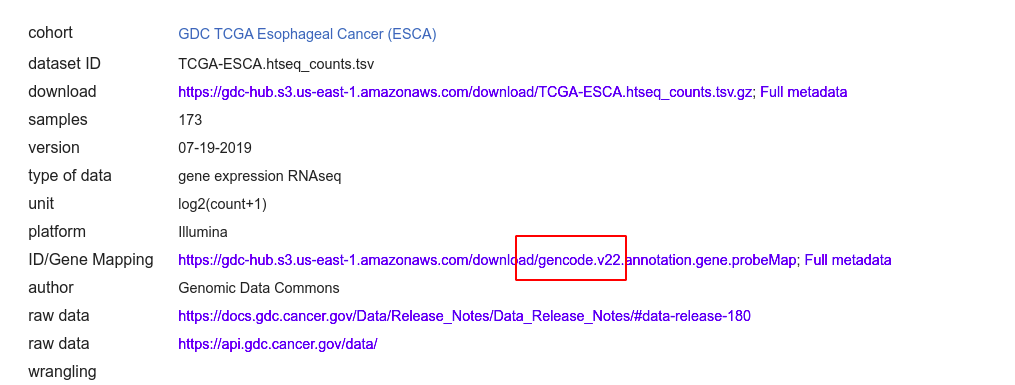
图片alt
recount2
- TCGA和GTEx数据合并的项目
broadinstitute
- R package RTCGA
- 下载
- 样本已经过筛选,数据标准化
- TCGA数据下载
gdac.broadinstitute.org_ESCA.Merge_rnaseqv2__illuminahiseq_rnaseqv2__unc_edu__Level_3__RSEM_genes_normalized__data.Level_3.2016012800.0.0.tar.gz
------------------解螺旋预后分析分析数据库------------------
Ualcan
- 轻松访问公开的癌症OMICS数据(TCGA,MET500和CPTAC)
- 允许用户识别生物标志物或对感兴趣的潜在基因进行计算机验证
- 提供描绘编码蛋白,miRNA编码和lincRNA编码基因的表达谱和患者生存信息
- 通过启动子甲基化评估基因表达的表观遗传调控
- 进行全癌基因表达分析
- 通过链接到HPRD,GeneCards,Pubmed,TargetScan,人类蛋白质图谱,DRUGBANK,Open Targets和GTEx,提供有关所选基因/靶标的其他信息
oncolnc
- 基因和生存相关性
kmplot
- 肿瘤生存分析
TRGAted(开源)
- TRGAted
- 肿瘤生存分析
TANRIC
- lncRNA在线分析
OncomiR
- miRNA在线分析
CCLE
- 癌症细胞百科全书
HPA
- 人类蛋白质数据库
TCPA
- 蛋白组学
------------------解螺旋预后分析分析数据库------------------
------------------解螺旋一站式分析数据库------------------
Oncomine
- 在没有目的基因的时候, 从数据库中筛选出候选分子作为今后的研究方向
- 在获得靶基因分子后, 利用数据库分析它们在肿瘤中的表达情况, 以及分子表达与临床生存时间, 预后相关性, 为研究目的提供更多依据.
- help
- 功能
- 基因差异表达分析
- 基因表达与临床相关性分析
- 生存分析
- 多基因共表达分析
Gepia
-
北大张泽民教授团队基于UCSC Xena计划的的数据库开发建造的数据库
-
可对TCGA基因表达谱数据进行动态分析和可视化的网站
-
功能
- 查询一个基因在一个或多个肿瘤的表达情况
- 查询某肿瘤中差异表达的基因
- 绘制某基因在某肿瘤中的生存曲线,或查询与某肿瘤最相关的100个基因
- 查询基因的共表达或相似基因
- 查询基因间相关性
- 生成可编辑用于发表的图片
MethHC
- MethHC 是一个人类泛癌甲基化数据库,侧重于人类疾病的 DNA 甲基化
- 在调控基因的转录过程时,DNA 的甲基化是很重要的一个环节。该数据库整合了诸如 DNA 甲基化、基因表达、microRNA 甲基化、microRNA 表达,以及来自 TCGA 的甲基化和基因表达的相关数据,能在探索基因调控机制上给予我们很大的帮助
LinkedOmics
- 包括来自所有32种TCGA癌症类型的多组学数据。它还包括由临床蛋白质组学肿瘤分析联合会(CPTAC)生成的基于质谱的蛋白质组学数据,用于TCGA乳腺,结肠直肠和卵巢肿瘤
CancerSEA
- R packag cancersea
- R packag CancerSEA
- 单细胞相关数据库
TSVDB(开源)
- 源码
- 有关可变剪切的
------------------解螺旋一站式分析数据库------------------