https://github.com/wangyang1749/yeast_data.git
酵母数据介绍
- 数据来自文章:In vivo targeting of de novo DNA methylation by histone modifications in yeast and mouse
- 该文章包括RAN-Seq数据、ChIP-Seq、DNA甲基化测序数据
- 背景:在酵母中没有DNA甲基化,该文章将DNA甲基化酶引入酵母。即转基因后,酵母基因组包含了基因甲基化,研究DNA甲基化影响表观遗传的状态。
- 本文介绍全基因组甲基化数据的处理流程
关于DNA甲基化
DNA甲基化是指在DNA甲基化转移酶的作用下,在基因组CpG二核苷酸的胞嘧啶5号碳位共价键结合一个甲基基团
图片alt
DNA Methylated 发生在胞嘧啶5号碳位

图片alt
在CpG岛,有甲基化基因表达可能被抑制,没有则可以正常表达

图片alt
DNA Methylation 的研究方法
全基因组DNA甲基化测序(Whole-genome bisulfite sequencing,WGBS)
其原理是用 Bisulfite 处理DNA序列,首先将基因组中未发生甲基化的 C 碱基转换成 U(T),从而与原本具有甲基化修饰的 碱基C区分开来,然后进行PCR扩增,结合高通量测序技术,适用于全基因组 范围内绘制单碱基分辨率的DNA 甲基化图谱。
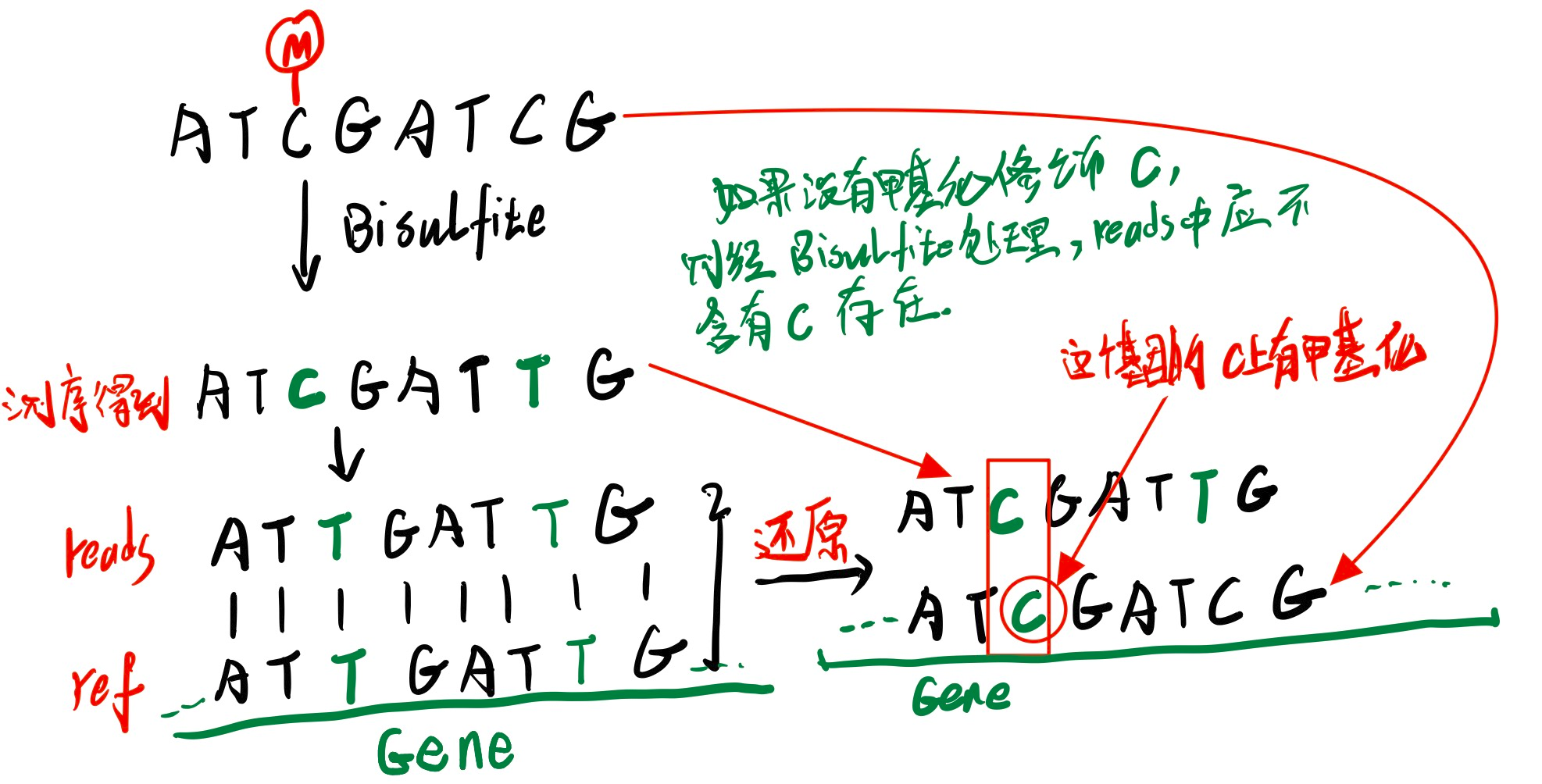
如下图所示,没有甲基化的Cytosine经过Bisulfite处理变成Thymine;有甲基化的Cytosine经过Bisulfite处理变成Cytosine。

图片alt

图片alt
3-letter alignment of Bisulfite-Seq reads
将参考基因组的C转换为T,同时也将reads的C也转换为T。此时相当于基因组只有3个碱基。比对之后在再将reads恢复到之前的序列。可以使用bismark进行比对。
图片alt