NCBI Taxonomy数据库中的nucl_gb.accession2taxid.gz、nucl_wgs.accession2taxid.gz和taxdump.tar.gz文件在功能和内容上有以下区别:
- nucl_gb.accession2taxid.gz
- 作用:存储GenBank中非WGS/TSA的核酸序列的accession号与taxid的映射关系。
- 内容:包含四列数据:accession(无版本号)、accession.version(带版本号的序列ID)、taxid(分类学ID)和gi(已弃用字段)。
- 应用场景:适用于通过BLAST比对到NT数据库后,将核酸序列的accession号转换为对应的物种分类ID。
- nucl_wgs.accession2taxid.gz
- 作用:存储全基因组测序(WGS)项目中核酸序列的accession号与taxid的映射关系。
- 与nucl_gb的区别:
- nucl_gb覆盖常规GenBank核酸序列(如单个基因或基因组片段),而nucl_wgs专门针对WGS项目提交的contig/scaffold序列。
- 两者在accession号格式和覆盖范围上存在差异,可能导致使用nucl_wgs提取子库时结果少于预期。
- taxdump.tar.gz
- 作用:提供NCBI分类学数据库的核心文件,包含物种分类层级结构和节点关系。
- 关键文件:
- nodes.dmp:记录分类学节点间的父子关系(如门→纲→目)。
- names.dmp:记录分类学ID与名称(学名、通用名、同义词等)的对应关系。
- 应用场景:与taxonkit等工具配合使用,将taxid转换为具体的物种分类信息(如界、门、纲等)。
三者的关联与典型使用流程
- 通过BLAST比对到NT数据库后,利用nucl_gb.accession2taxid.gz或nucl_wgs.accession2taxid.gz获取序列对应的taxid。
- 结合taxdump.tar.gz中的分类学信息,将taxid转换为物种分类层级(如Bacteria;Proteobacteria;...)。
总结表格
| 文件 | 内容范围 | 核心用途 | 依赖工具/场景 |
|---|---|---|---|
| nucl_gb.accession2taxid.gz | 非WGS核酸序列 | 序列accession转taxid | BLAST结果注释 |
| nucl_wgs.accession2taxid.gz | WGS项目核酸序列 | WGS序列accession转taxid | 子库提取 |
| taxdump.tar.gz | 分类学节点与名称 | taxid转物种分类层级 | TaxonKit等工具 |
通过以上文件组合,可完成从序列比对到物种注释的全流程分析。
从refSeq下载物种基因组

从RefSeq genomes FTP可以下载refSeq的物种基因组。
下载refseq所有细菌基因组
wget https://ftp.ncbi.nlm.nih.gov/genomes/refseq/bacteria/assembly_summary.txt
$ cat assembly_summary.txt | wc -l
# 417112
$ du -sh assembly_summary.txt
# 177M assembly_summary.txt
taxonomy中统计的细菌数
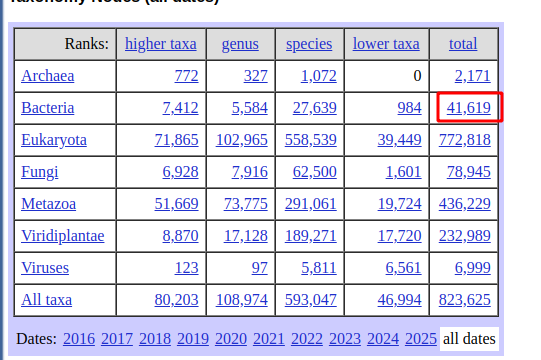
通过Linux命令行FTP查看NCBI细菌基因组目录的步骤
ftp ftp.ncbi.nlm.nih.gov
输入用户名 anonymous,密码留空或填写邮箱地址(如 user@example.com)完成匿名登录
ftp> cd genomes/refseq/bacteria