- 概念
- 目的
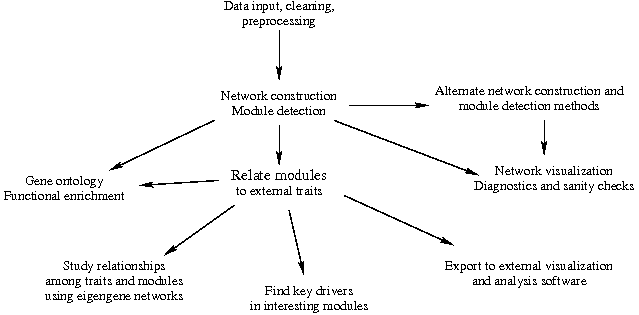
- 步骤
- Data input and cleaning
- Automatic, one-step network construction and module detection
- 与外部临床特征相关的模块,并识别重要基因
- Step-by-step network construction and module detection
- 处理大型数据集:分块网络构建和模块检测
- 将网络分析与功能注释和基因本体等其他数据连接起来
- 使用WGCNA函数网络可视化
- 将网络导出到外部软件
- Can WGCNA be used to analyze RNA-Seq data?
- 参考
概念
WGCNA(Weighted Correlation Network Analysis),加权基因共表达网络分析,与共表达分析不同。
WGCNA是为了芯片数据开发的,能否直接用于测序数据?
测序数据是泊松分布,而是负二项分布
权重=correlation
WGCNA假设,表达相关性高的基因必然功能在同一通路上或有相互作用
目的
寻找具有协同表达(共表达)的基因组成的网络模块,探索基因网络模块与研究的表性性状之间的联系,寻找外部信息相关的hub基因,为下一步研究实验设计提供指导
- 根据雌性小鼠肝脏表达数据的网络分析,寻找与体重相关的模块
- WGCNA不是对样本聚类,而是对变量(基因)聚类
步骤
- 定义gene之间距离的计算方式
- 根据基因之间的距离生成聚类结果
- 设定一个cutoff,将聚类结果划分为不同的module(共表达模块),或者说将网络分割成局部子图
- 在module中选取hub基因
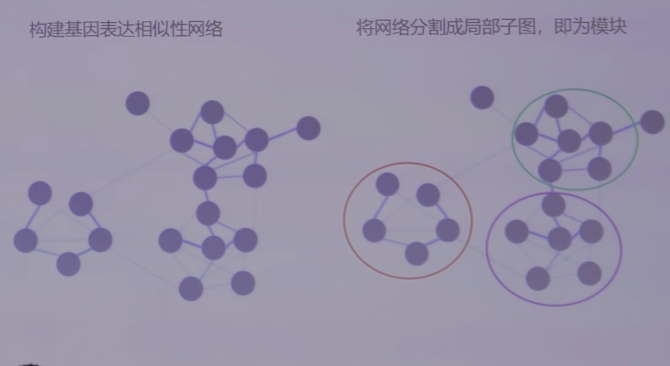
图片alt
构建基因表达相似性网络,每一个节点是一个基因或者蛋白质,节点有显著的相关性,通过边连接,边的权重是相关系数的绝对值,之后将网络分割为局部的子图(模块)

图片alt
四个基因两两相关性的网络,边的权重为二元相关性

图片alt
对于一个基因有两种指标,连通度和拓扑重叠度

图片alt
图片alt
Data input and cleaning
样本聚类
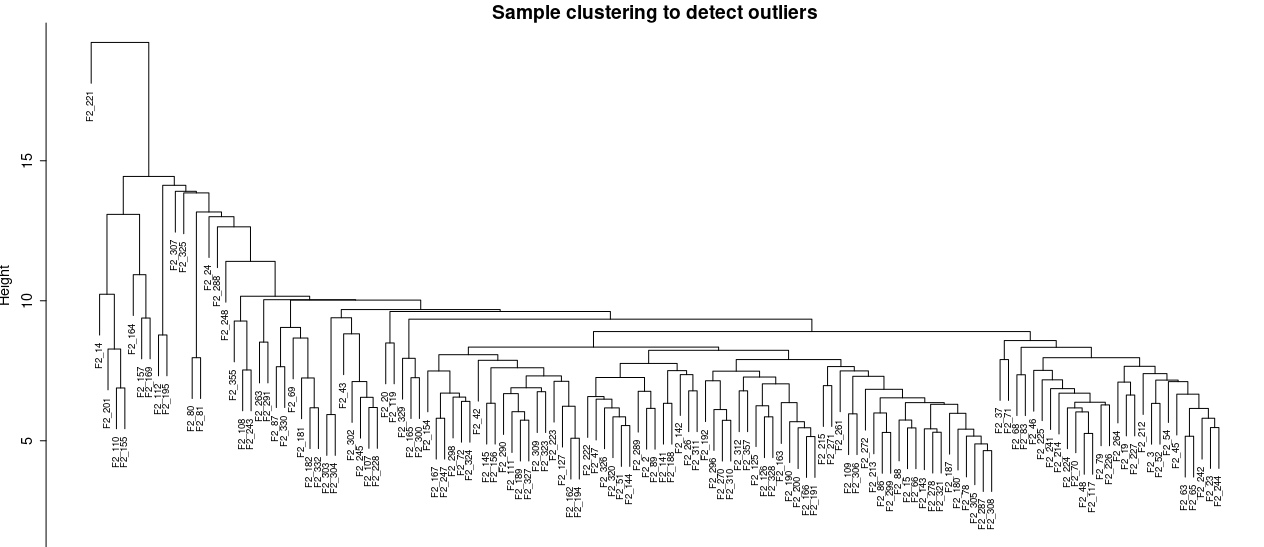
图片alt
F2_221为异常样本
选择一个高度切割,剔除异常样本

图片alt
临床特征与样本树状图的关系可视化

图片alt
Automatic, one-step network construction and module detection
软阈值功率(soft-thresholding power)的选择:网络拓扑分析
Choosing the soft-thresholding power: analysis of network topology

图片alt
不同软阈值功率(soft-thresholding powers)下的网络拓扑分析。左面板显示了作为软阈值功率( soft-thresholding power)(x轴)函数的无标度拟合指数( scale-free fit index)(y轴)。右面板显示作为软阈值功率(soft-thresholding power)(x轴)函数的平均连通性(the mean connectivity)(度,y轴)
如图所示,选择power 6,这是无标度拓扑拟合指数曲线在达到较高值(在本例中,约为0.90)时变平的最低幂
一步网络构建与模块检测
One-step network construction and module detection

图片alt
基于拓扑重叠的差异性基因聚类树状图,以及指定的模块颜色
与外部临床特征相关的模块,并识别重要基因
Relating modules to external clinical traits and identifying important genes
确定临床特征显著相关的模块

图片alt
Step-by-step network construction and module detection
处理大型数据集:分块网络构建和模块检测
Dealing with large datasets: block-wise network construction and module detection
将网络分析与功能注释和基因本体等其他数据连接起来
Interfacing network analysis with other data such as functional annotation and gene ontology
使用WGCNA函数网络可视化
Network visualization using WGCNA functions
将网络导出到外部软件
Export of networks to external software
Can WGCNA be used to analyze RNA-Seq data?
我们建议进行方差稳定变换(variance-stabilizing transformation),例如,包DESeq2实现了我们发现很有用的函数varianceStabilizingTransformation,但也可以从标准化计数(或RPKM/FPKM数据)开始,并使用log2(x+1)对它们进行 log2(x+1)转换。对于高度表达的功能,完全方差稳定和简单对数变换之间的差异很小。
无论是使用RPKM、FPKM还是简单的标准化计数,只要所有样本都以相同的方式处理,WGCNA分析就不会有太大差异。如果想要比较基因a和基因B的表达,这些标准化方法会产生很大的不同;但WGCNA计算的相关性在基因尺度上没有区别。(当然,样本的比例因子确实如此,所以样本确实需要标准化。)
如果数据来自不同批次,我们建议检查批次效应,如果需要,进行调整。我们使用ComBat来消除批量效应,但其他方法也应该有效。最后,我们通常检查分位数散点图,以确保样本之间没有系统性偏移;如果样本分位数显示相关性(通常如此),可以使用分位数归一化来消除这种影响。
共表达分析
https://translatome.net/Resources/2014ben/Bioinfo_jnu.html
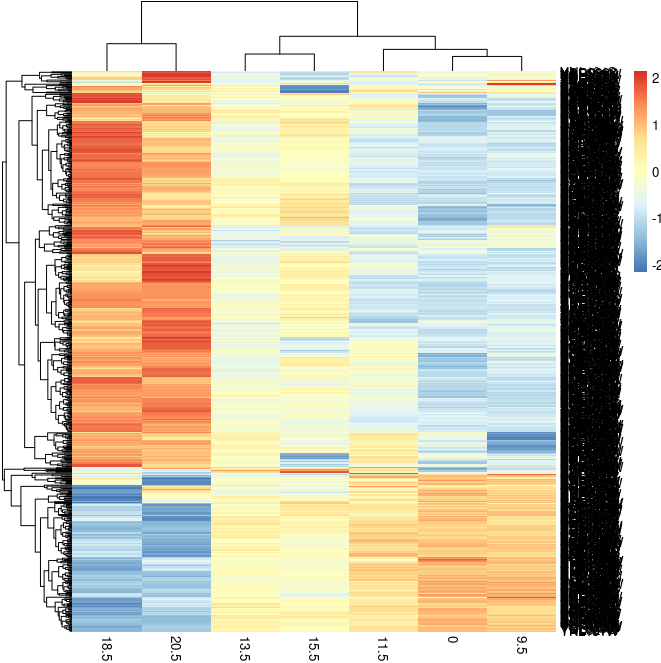
图片alt
pheatmap(yeastdata,scale="row")
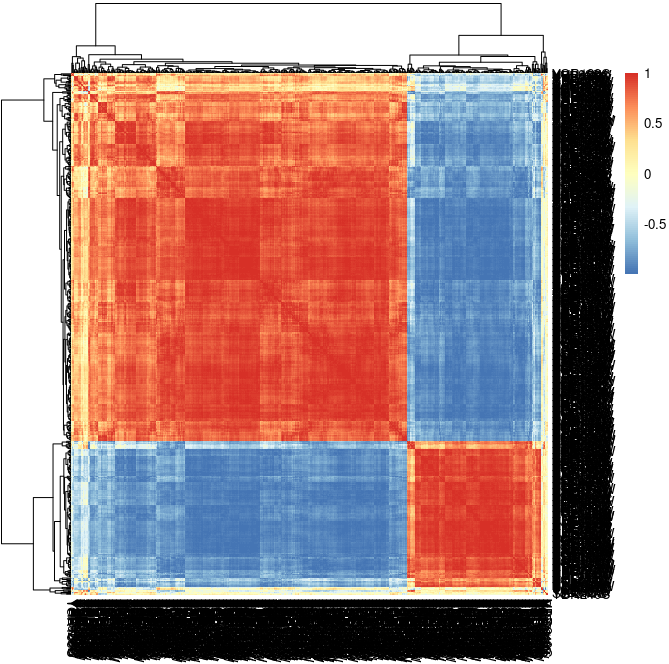
图片alt
pheatmap(cor(t(yeastdata)))